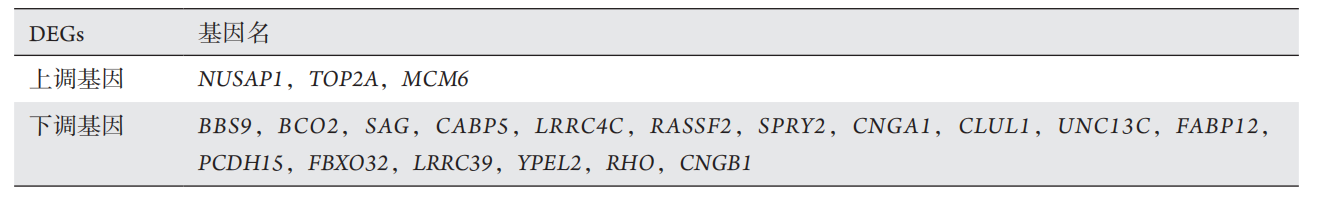
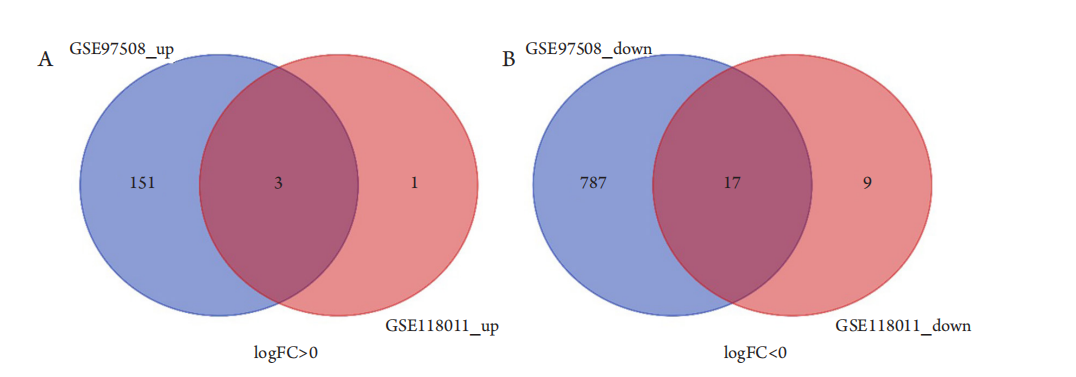
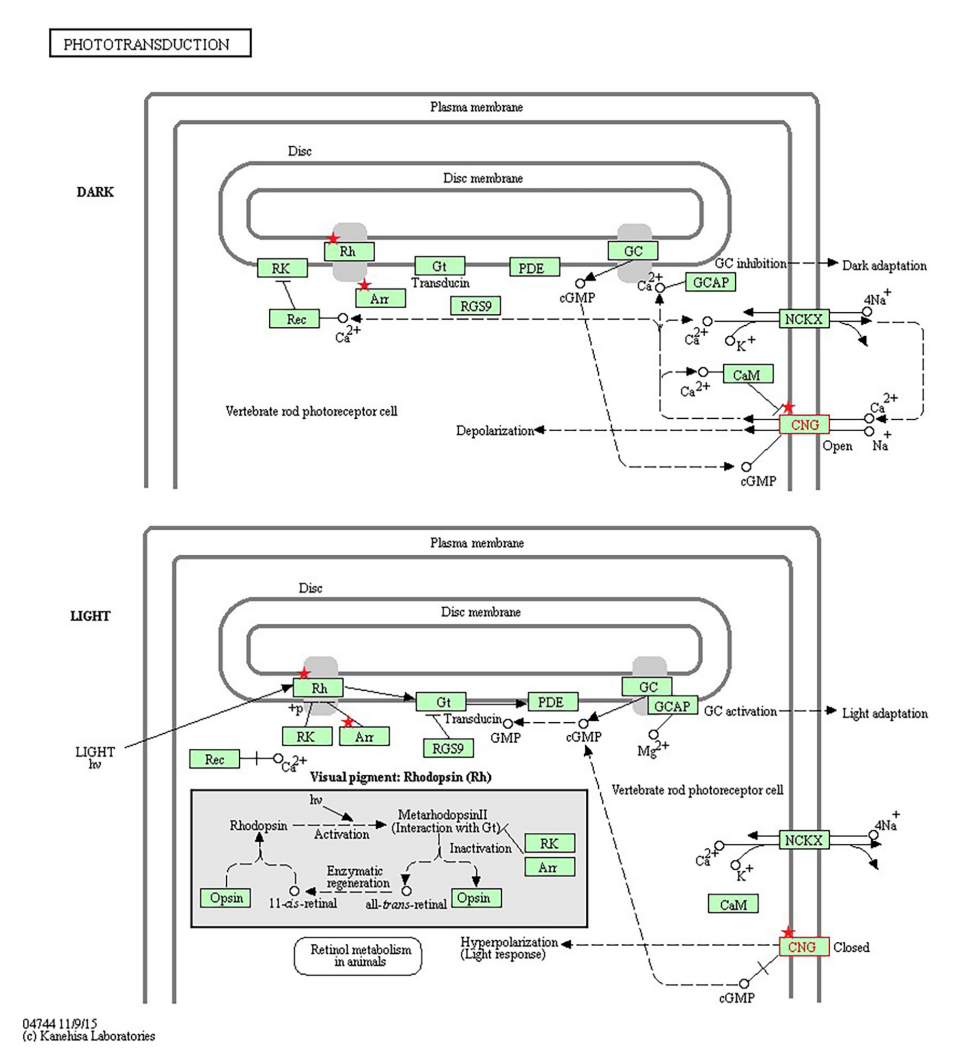
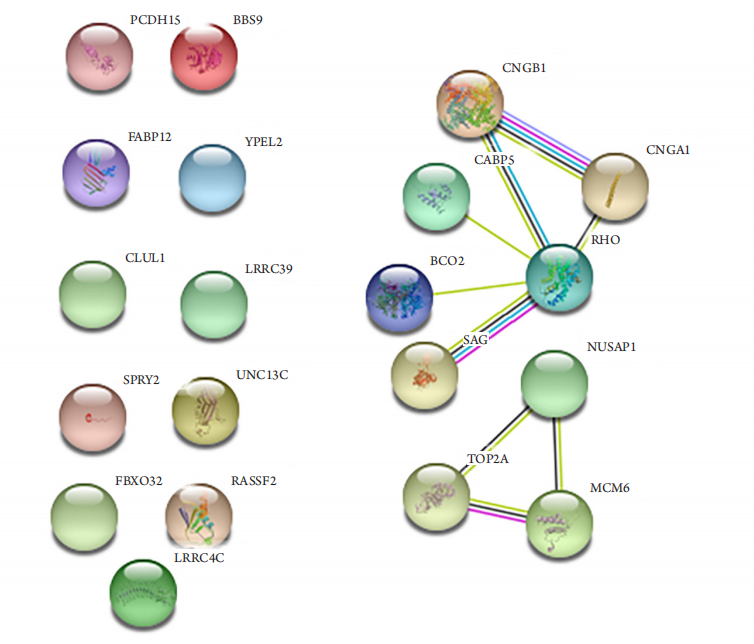
1、Dimaras H, Corson TW. Retinoblastoma, the visible CNS tumor: A
review[ J]. J Neurosci Res, 2019, 97(1): 29-44.Dimaras H, Corson TW. Retinoblastoma, the visible CNS tumor: A
review[ J]. J Neurosci Res, 2019, 97(1): 29-44.
2、Zhang J, Benavent AC, McEcoy J, et al. A novel retinoblastoma therapy from
genomic and epigenetic analyses[J]. Nature, 2012, 481(7381): 329-334.Zhang J, Benavent AC, McEcoy J, et al. A novel retinoblastoma therapy from
genomic and epigenetic analyses[J]. Nature, 2012, 481(7381): 329-334.
3、Bert V, Nickolas P, Velculescu VE, et al. Cancer genome landscapes[ J].
Science, 2013, 339(6127): 1546-1558.Bert V, Nickolas P, Velculescu VE, et al. Cancer genome landscapes[ J].
Science, 2013, 339(6127): 1546-1558.
4、Hudson LE, Mendoza P, Hudson WH, et al. Distinct gene expression
profiles define anaplastic grade in retinoblastoma[ J]. Am J Pathol,
2018, 188(10): 2328-2338.Hudson LE, Mendoza P, Hudson WH, et al. Distinct gene expression
profiles define anaplastic grade in retinoblastoma[ J]. Am J Pathol,
2018, 188(10): 2328-2338.
5、Ashburner M, Ball CA, Blake JA, et al. Gene ontology: tool for the
unification of biology. The Gene Ontology Consortium[ J]. Nat Genet,
2000, 25(1): 25-29.Ashburner M, Ball CA, Blake JA, et al. Gene ontology: tool for the
unification of biology. The Gene Ontology Consortium[ J]. Nat Genet,
2000, 25(1): 25-29.
6、Kanehisa M, Goto S. KEGG: Kyoto encyclopedia of genes and
genomes[ J]. Nucleic Acids Resh, 2000, 28(1): 27-30.Kanehisa M, Goto S. KEGG: Kyoto encyclopedia of genes and
genomes[ J]. Nucleic Acids Resh, 2000, 28(1): 27-30.
7、Szklarczyk D, Franceschini A, Wyder S, et al. STRING v10: protein�protein interaction networks, integrated over the tree of life[ J]. Nucleic
Acids Res, 2015, 43: D447-D452.Szklarczyk D, Franceschini A, Wyder S, et al. STRING v10: protein�protein interaction networks, integrated over the tree of life[ J]. Nucleic
Acids Res, 2015, 43: D447-D452.
8、Sameh E, Soliman HR , Cheng Y, et al. Genetics and molecular
diagnostics in retinoblastoma—an update[ J]. Asia Pac J Ophthalmol,
2017, 6(2): 197-207.Sameh E, Soliman HR , Cheng Y, et al. Genetics and molecular
diagnostics in retinoblastoma—an update[ J]. Asia Pac J Ophthalmol,
2017, 6(2): 197-207.
9、Donoso L A , Hamm H, Dietzschold B, et al. Rhodopsin and
Retinoblastoma: a monoclonal antibody histopathologic study[ J].
Arch Ophthalmol, 1986, 104(1): 111-113.Donoso L A , Hamm H, Dietzschold B, et al. Rhodopsin and
Retinoblastoma: a monoclonal antibody histopathologic study[ J].
Arch Ophthalmol, 1986, 104(1): 111-113.
10、Hurwitz RL, Bogenmann E, Font RL, et al. Expression of the functional
cone phototransduction cascade in retinoblastoma[ J]. J Clin Invest,
1990, 85(6): 1872-1878.Hurwitz RL, Bogenmann E, Font RL, et al. Expression of the functional
cone phototransduction cascade in retinoblastoma[ J]. J Clin Invest,
1990, 85(6): 1872-1878.